El XGboost (Extra Gradient boosting) parte del árbol de decisiones que se implementa en minería de datos para clasificar o pronosticar sobre una variable objetivo (y), a través, del aprendizaje automático que se realiza sobre un set de datos, utilizando varios clasificadores débiles que en este caso, como ya mencionamos, son nuestros árboles de decisiones, pero potenciando los resultados de estos, debido al procesamiento secuencial de la data con una función de pérdida o coste, la cual, minimiza el error iteración tras iteración, haciéndolo de esta manera, un pronosticador fuerte.
Sin embargo, como siempre esto va a depender del nivel de ajuste de los parámetros que utilicemos en la función: xgb, ya que, determinará los resultados del modelo.
Así pues, indicamos las principales ventajas de utilizar este método, en nuestros proyectos:
- Trabaja sobre grandes bases de datos con múltiples variables, tanto categóricas como numéricas.
- Funciona en ambientes de python como en R.
- Admite missing values, o valores perdidos en medio del procesamiento del algoritmo.
No obstante, presenta las siguientes desventajas:
- Utiliza grandes cantidades de memoria RAM, por lo que, es recomendable procesar el algoritmo determinando previamente mediante una validación cruzada, las variables que más aportan información a la estructuración del modelo,
- En efecto, se recomienda equipos de más de 8GB para correr bases de datos extensas si se quiere trabajar con todas la variables.
- Se debe ajustar correctamente los parámetros para minimizar el error de precisión.
# El método Xgboost utiliza las siguientes paqueterias: library(xgboost); library(magrittr); library(dplyr); library(Matrix); library(sqldf); library(gsubfn); library(proto); library(RSQLite); library(plyr)
#Leemos el archivo maestro, en este caso, la base de datos interna: Iris (150 filas, 5 columnas) data(iris) datos=iris #Convertimos la variable: Species, de categórica a numérica. datos[,5]=as.numeric(factor(datos[,5]))-1 #Seleccionamos la data de aprendizaje (75%) y la data de prueba (25%). muestra=sample(nrow(datos), (nrow(datos)*0.75)) taprendizaje=datos[muestra,] ttesting=datos[-muestra,]
# Creación de la Matrices para entrenamiento y prueba con la librería: XgBoost. #Aceptación de registros con campos de valores perdidos (opcional). options(na.action='na.pass') #Construcción de la matriz de datos: Sparse (*), a ser procesada por el algorimo. trainm <- sparse.model.matrix(Species ~.-1, data = taprendizaje) #* #* se suprime el campo de intercepto que crea la función:sparse, con el -1. #Variable objetivo o etiqueta. train_label <- taprendizaje[,"Species"] #Se crea la matriz de la data de entrenamiento con la libreria: xgboost, donde se introduce la matrix sparse anterior (*). train_matrix <- xgb.DMatrix(data =as.matrix(trainm), label= train_label) #Se repite el proceso anterior, pero esta vez con el set de pruebas. testm <- sparse.model.matrix(Species~.-1, data = ttesting) test_label <- ttesting[,"Species"] test_matrix <- xgb.DMatrix(data = as.matrix(testm), label = test_label)
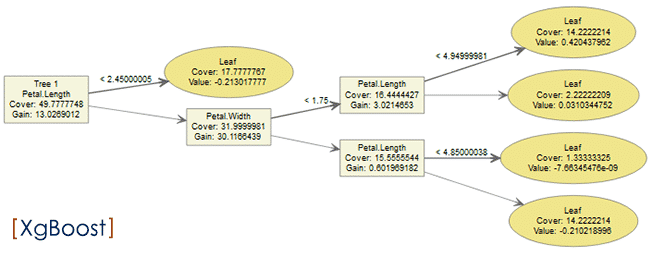
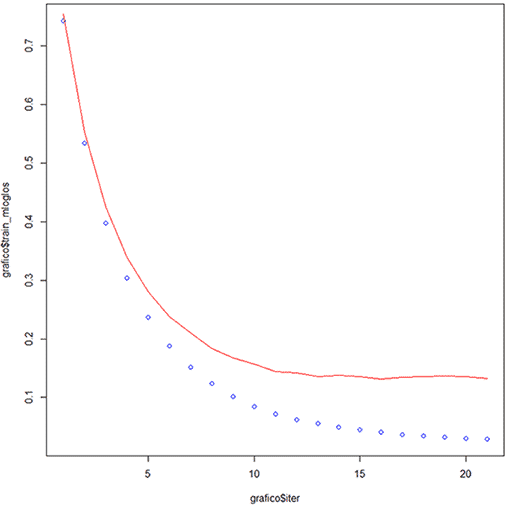
#Ejecutamos el algoritmo del XGBOOST con la matriz sparse de entrenamiento, con los siguientes parámetros: xgb <- xgb.train( objective = "multi:softprob", eval_metric = "mlogloss", data = train_matrix, max_depth = 6, eta = 0.3, nrounds = 21, missing =NA, num_class =3, watchlist = list(train=train_matrix, test=test_matrix) ) #Verificamos los elementos de la función xgb.train xgb # Gráfico del test de entrenamiento. grafico<- data.frame(xgb$evaluation_log) #Curva del dataset: train_matrix, de la variable:train_mloglos. plot(grafico$iter, grafico$train_mloglos, col = 'blue') #Curva del dataset: test_matrix, de la variable:test_mlogloss. lines(grafico$iter, grafico$test_mlogloss, col = 'red')
#¡ Importante ! Sobre el gráfico que se genera por la sentencia: grafico=data.frame(xgb$evaluation_log) plot(grafico$iter, grafico$train_mloglos, col = 'blue') lines(grafico$iter, grafico$test_mlogloss, col = 'red')Es fundamental hacer notar como desciende el error: mlogloss, durante la fase de entrenamiento: curva azul, a medida que se desarrollan las 21 iteraciones, así como, también sucede los mismo, en la fase de prueba: curva roja. La determinación del número de iteraciones proviene de realizar una validación cruzada a dónde se decide el número exacto de nrounds (iteraciones), porque este método permite de forma óptima obtener la coordenada dónde la función tiene su mínimo valor, que en este caso, indicará el menor error.
Otro comentario de relevada importancia es que los valores de los hiperparámetros: objective, eval_metric, data, max_depth, eta, nrounds, missing, num_class y watchlist , corresponden únicamente a las finalidades de este ejercicio, por consiguiente, existe una enorme probabilidad de que no funcionen correctamente en otro ejercicio, sin embargo, todo dependerá del mismo.
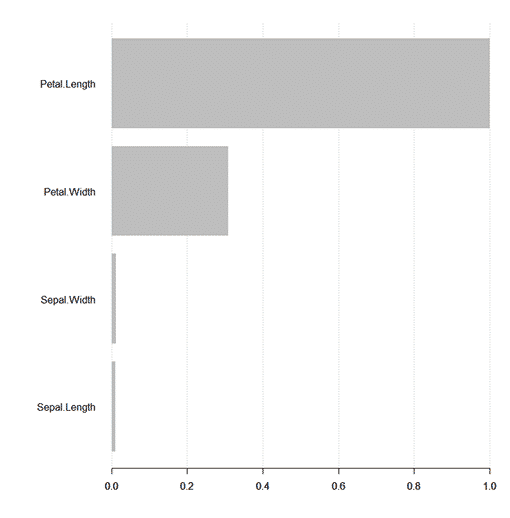
# Variables de Importancia. imp=xgb.importance(colnames(train_matrix), model = xgb) print(imp) #Gráfico de la importancia de las variables. importance=xgb.importance(feature_names = colnames(trainm),model= xgb) #Gráfico de la importancia de las variables. #Todas las Variables. xgb.plot.importance(importance_matrix = importance,rel_to_first = T)
#¡ Importante ! Sobre el gráfico que se genera por la sentencia: xgb.plot.importance(importance_matrix = importance,rel_to_first = T)Se puede observar que las variables que más aportan información en la clasificación son las siguientes por orden de importancia: 1. Petal.Length, 2. Petal.Width, 3. Sepal.Width y, 4.Sepal.Length, por lo tanto, podemos observar las variables en las que tenemos que enfocarnos para hallar nuestras soluciones.
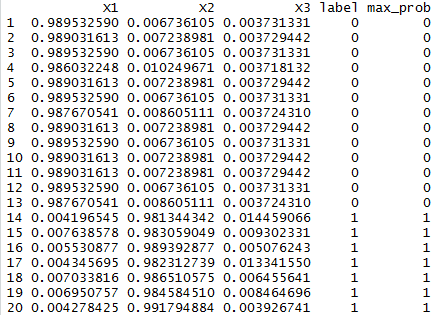
# Pronóstico sobre el test de prueba. #Longitud de la etiqueta: train_label. nc=length(unique(train_label)) #Valores pronósticados. p=predict(xgb, newdata = test_matrix) head(p) #Valores pronósticados en formato: 0 y 1, por la variable: max_prob. pred % t() %>% data.frame() %>% mutate(label = test_label, max_prob = max.col(., "last")-1 ) head(pred,20)
#¡ Importante ! Sobre el output que se genera por la sentencia: head(pred,20)Es notable que aparecen cinco columnas: X1, X2, X3, label y, maxprob, donde:
X1, X2 y X3: Indican la distribución de la probabilidad de cada opción que puede tomar la variable: Species, por ende, corresponden a los tipos de iris: Setosa, Versicolor y Virginica, respectivamente.
Label: Es la decisión entre: 0,1 o 2, es decir, Setosa, Versicolor o Virginica, por la cual, el algoritmo de XgBoost, según la probabilidad de las variables: X1,X2 y X3, ha tomado.
Max_prob: Es la decisión final tomada por el algoritmo XgBoost con la máxima probabilidad, por ende, está asociada con el valor de la variable Label.
#Matriz de Confusión. Matriz_Confusion=table(Prediction = pred$max_prob, Actual = pred$label) Matriz_Confusion #Precisión. Precision=( sum(diag(Matriz_Confusion) ))/sum(Matriz_Confusion) Precision #Error. Error=1-Precision Error
#¡ Importante ! Sobre los outputs que se generan por las sentencias: Matriz_Confusion Precision ErrorComparando los resultados obtenidos entre la implementación de los algoritmos de Kmeans y XgBoost, se puede indicar que este último es más exacto con una precisión de +/- 94.73%, mientras, que el primero es de 89.3%, por lo cual, se puede indicar que con el XgBoost se obtienen más casos pronosticados correctamente.
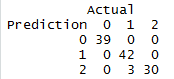




Hola,
Muy interesante el artículo y muy explicativo. Creo que faltan más artículos como este para que la gente se anime mucho más a utilizar los algoritmos de Machine Learning.
Te animo a que sigas escribiendo.
Un saludo,
Gracias Mario, sin duda alguna, enseñar a los iberoamericanos esta tecnología, sin duda, alguna hará posible una mejor iberoamérica.
Gran artículo;
Me gustaría usar este algoritmo para intentar predecir posibles cuellos de botella en ciertos contadores de rendimiento de mis servidores;
Por donde podría empezar ?
Muchas gracias de antemano;
Hola genial se entiende muy bien intente adaptarlo a otro dataset que contiene otra variable a predecir su valor es 0 , 1
Se presenta problema al correr el ultimo paso: #Valores pronósticados en formato: 0 y 1, por la variable: max_prob.
Recibo como error Error:
«in t.default() : argument «x» is missing, with no default»
y no pude encontrar solucion, sabran orientarme que podria ser. Gracias!
Hola Sergio, tengo el mismo inconveniente, lo pudiste solucionar?. Gracias de antemano.
Excelente, solo podrian revisar la parte final de la prediccion, me genera un error aqui :
#Valores pronósticados en formato: 0 y 1, por la variable: max_prob.
pred %
t() %>%
data.frame() %>%
mutate(label = test_label, max_prob = max.col(., «last»)-1 )
head(pred,20)